
On ne compte plus les études associant la dysbiose intestinale ou de « mauvaises bactéries » à l’incidence de maladies, physiques comme mentales. Sans compter les recherches qui documentent les axes intestin-cerveau ou plus récemment intestin-peau…Cependant, l’analyse de la composition génétique de ces milliards de bactéries dans l'intestin humain et des liens pouvant relier certains groupes de gènes bactériens, ou «signatures génétiques», aux maladies relevait jusque-là de l’impossible, en regard des volumes de données à traiter. Ces travaux d’une équipe de l’École de médecine de Harvard, publiés dans la revue Nature Communications, montrent, qu’avec l’aide des sciences computationnelles, il devient aujourd’hui possible de relier l’énorme quantité de données génétiques des bactéries de l'intestin humain, à plusieurs maladies humaines. En pratique, très bientôt, l’analyse d’un simple échantillon fécal pourrait nous permettre de faire un check up complet.
« Nous ne sommes jamais vraiment seuls, même pas dans notre propre corps car nous hébergeons ces milliards de bactéries, champignons, virus et autres micro-organismes qui composent le microbiome humain », relèvent les chercheurs de la Harvard Medical School (HMS) et du Joslin Diabetes Center (Massachusetts). Leur objectif, analyser ce mélange de bactéries résidentes pour diagnostiquer tout un spectre de conditions, allant de l'obésité à la sclérose en plaques. Avec très prochainement, une application diagnostique toute simple, un test d’échantillon du microbiome.
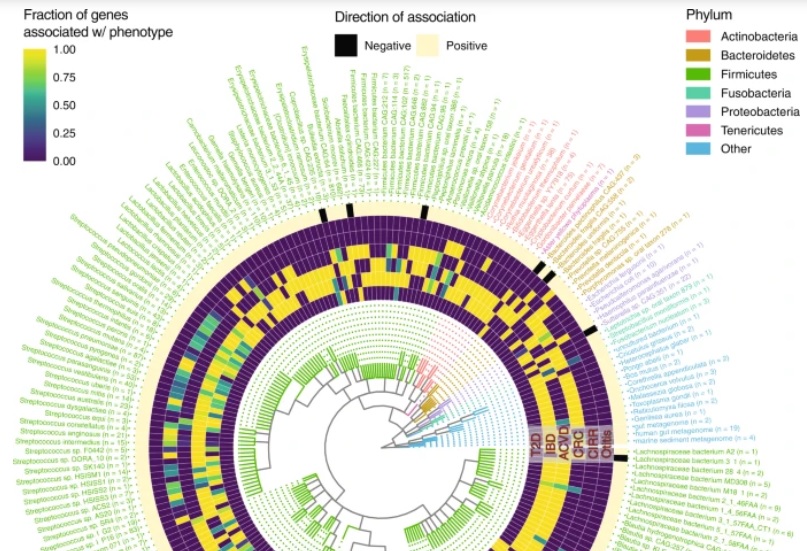
Des groupes de gènes bactériens associés aux différentes maladies
Les chercheurs ont commencé par collecter des données sur le microbiome de 13 groupes de patients à travers l’analyse de plus de 2.500 échantillons fécaux. Ensuite, ils ont analysé les données pour identifier les liens entre 7 maladies et des millions d'espèces microbiennes, de voies métaboliques microbiennes et de gènes microbiens. A l’aide de plusieurs modèles statistiques, les scientifiques ont pu identifier les caractéristiques du microbiome les plus puissamment associées à la maladie. De toutes les caractéristiques microbiennes – espèces, voies et gènes – les gènes microbiens présentent le pouvoir prédictif le plus fort. En d'autres termes, les groupes de gènes bactériens, ou signatures génétiques, plutôt que la simple présence de certaines familles bactériennes, sont plus étroitement liés à la présence d'une condition/maladie donnée.
Quelques exemples de résultats « concrets » : ici, les chercheurs parviennent à relier des ensembles de gènes bactériens à la présence d'une maladie coronarienne, d'une cirrhose du foie, d'une maladie inflammatoire chronique de l'intestin (MICI), d'un cancer du côlon et d'un diabète de type 2. 3 de ces conditions (la maladie coronarienne, la MICI et la cirrhose du foie) apparaissent partager de nombreux gènes bactériens. En d'autres termes, les personnes dont le microbiome comprend ces gènes bactériens sont plus susceptibles d'avoir une ou plusieurs de ces 3 conditions.
L'analyse d'un seul échantillon fécal pour un check up complet : ces travaux représentent une avancée significative dans la compréhension de la relation entre microbiote intestinal et maladies : ces premières données qui restent encore à élargir avec d’autres relations entre gènes bactériens et maladies, vont bientôt permettre le développement de nouveaux outils diagnostiques permettant de prédire et/ou d’évaluer le risque d'une personne pour toute une gamme de maladies, relève le chercheur Braden Tierney, du programme de sciences biologiques et biomédicales de la HMS :
«Nous identifions des marqueurs génétiques qui vont conduire à des tests, ou à un seul test, permettant d’identifier les associations avec toute une série de conditions médicales ».
Bactéries spectatrices ou responsables ? Il « reste en effet à savoir » si ces bactéries identifiées comme associées, sont également impliquées dans le développement de la maladie ou ne sont que de simples « spectatrices » dans ce processus. Mais au-delà des perspectives de test prédictif unique, ces travaux nous enseignent que les signatures génétiques des microbes comptent plus que la présence d'une famille bactérienne donnée pour présager un risque.
L’analyse des « big data » via les sciences computationnelles permet aujourd’hui de travailler à partir du grand nombre de gènes microbiens qui résident dans le corps humain, et de démêler la complexité de l'interaction entre les maladies humaines et le microbiome humain.
« Il devient possible de générer des hypothèses puis des conclusions à partir d'une énorme quantité de données ».
Source: Nature Communications 18 May 2021 DOI : 10.1038/s41467-021-23029-8 Gene-level metagenomic architectures across diseases yield high-resolution microbiome diagnostic indicators
Plus sur Microbiote Blog
